"Stille Mutationen“ können Krebszellen verändern
Direktzugriff
Artikelaktionen
Genveränderungen im Protein-Bauplan galten bislang als irrelevant, wenn trotzdem die gleichen Protein-Bausteine hergestellt werden. Nun zeigen Freiburger Forscher, dass diese Mutationen durchaus die Proteinaktivität in Krebszellen verändern können.
Bislang dachte man, dass Änderungen im Erbgut ohne Folgen bleiben, wenn es dadurch nicht zu einem Austausch von Proteinbausteinen kommt. Man sprach dann von „stillen Mutationen“. Nun zeigen Freiburger Forscherinnen und Forscher, dass derartige Veränderungen aber durchaus die Zellaktivität verändern können. Das Team um Prof. Dr. Sven Diederichs, der die Abteilung für onkologische Forschung der Klinik für Thoraxchirurgie am Universitätsklinikum Freiburg und die Abteilung RNA Biology and Cancer des Deutschen Krebsforschungszentrums (DKFZ) leitet, untersuchte 88 Tumorarten und mehr als 650.000 Mutationen. „Am Beispiel des bedeutenden Krebs-Gens KRAS konnten wir zeigen, dass eine vermeintlich stille Mutation die Protein-Bildung verringert. Stille Mutationen sind also gar nicht so still“, so Diederichs. Die Forscher entwickelten eine benutzerfreundliche Datenbank, SynMICdb, die bekannte stille Mutationen listet und so anderen Wissenschaftlern entsprechende Auswertungen deutlich vereinfacht. Ihre Datenbank und konkrete Ergebnisse stellen die Forscher am 12. Juni 2019 im Fachmagazin Nature Communications vor.
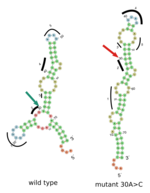
Eine solche Fehlfaltung der mRNA konnten Bioinformatiker der Albert-Ludwigs-Universität Freiburg auch am Computer simulieren. „Wir konnten zeigen, dass sich die räumlichen Eigenschaften auch bei eigentlich stillen Mutationen gravierend verändern können“, sagt Prof. Dr. Rolf Backofen vom Institut für Informatik der Albert-Ludwigs-Universität Freiburg.
Der genetische Code des Lebens hat Redundanz, dass die meisten Aminosäuren durch mehrere Codons kodiert werden. Milad Miladi, Doktorand am Lehrstuhl für Bioinformatik, erklärt diese Redundanz am Beispiel von Computerprozessoren. "Die Protein herstellende Maschinerie übersetzt die 64 verschiedenen RNA-Tripletts (biologische Opcodes) auf nur etwa 21 Aminosäuren und einige wenige Produktionsanweisungen. Wenn der Code des Lebens ein Prozessor Maschinencode gewesen wäre, dann ist seine Redundanz wie eine Rechnerarchitektur mit mehreren Befehlscodes für die gleiche arithmetische Operation. Wir haben gezeigt, dass auch die synonymen Verzerrungen Auswirkungen auf die Zellen haben. Der Effekt ist vergleichbar mit dem Umschalten zwischen zwei logisch identischen Computerprogrammen, die unterschiedliche Speicherfragmentierung und Ladezeiten haben und damit das Verhalten der Programme verändern."
„Derartige Veränderungen könnten künftig in der Krebstherapie eine wichtige Rolle spielen, etwa weil entscheidende Proteine stärker oder schwächer gebildet werden. Davor müssen aber die Folgen synonymer Mutationen deutlich besser erforscht werden. Das ist nun dank unserer Online-Datenbank auch für Wissenschaftler möglich, die keine vertieften bioinformatischen Kenntnisse mitbringen“, sagt Diederichs.
Originalveröffentlichung:
Sharma, Y., Miladi, M., Dukare, S., Boulay, K., Caudron-Herger, M., Groß, M., Backofen, R. and Diederichs, S., 2019. A pan-cancer analysis of synonymous mutations. Nature communications, 10 no. 1 pp. 2569, 2019.
Kontakt:
Prof. Dr. Sven Diederichs
Abteilung für onkologische Forschung
Klinik für Thoraxchirurgie
Universitätsklinikum Freiburg
Telefon: 0761 270-77571
sven.diederichs@uniklinik-freiburg.de
Prof. Dr. Rolf Backofen
Lehrstuhl für Bioinformatik
Institut für Informatik
Albert-Ludwigs-Universität
Telefon: 0761 203-7461
backofen@informatik.uni-freiburg.de
Fußzeile
Benutzerspezifische Werkzeuge